基因敲除的sgRNA设计原理
基因敲除是指使生物体基因组中的特定基因失活或失效的过程。通常是通过在靶基因中引入突变来实现的,使其失去功能。进行基因敲除的方法,包括同源重组、RNA干扰(RNAi)和CRISPR/Cas9。同源重组即通过引入修饰的DNA片段,用无功能或被破坏的版本替换靶基因;RNAi利用小RNA分子来沉默或降解靶基因的信使RNA(mRNA),阻止其翻译为蛋白质;CRISPR/Cas9是一种革命性的基因编辑工具,它使用引导RNA(sgRNA)和Cas9酶在基因组的特定位置引入靶向DNA断裂,导致基因破坏。
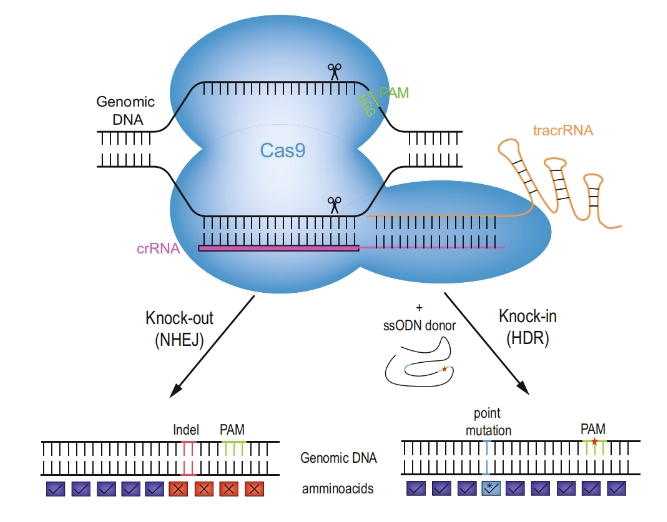
基因敲除的sgRNA设计原理
在 CRISPR-Cas9 系统中,sgRNA(small guide RNA,sgRNA)通过碱基互补配对原则引导 Cas9 蛋白酶 至基因组特定的靶序列上,Cas9 蛋白与 PAM 序列结合后便可对靶位点进行切割,形成 DNA 双链切口。sgRNA 设计的合理与否,对于 CRISPR-Cas9 编辑系统的切割效率,甚至后能否得到 KO 细胞具有重要影响。
目前虽然有诸多高效快速的 gRNA 设计工具,但我们仍需了解基本的设计原则才能更好地做出判断。sgRNA 的设计需要遵循以下原则:
(1) 不影响其他基因,尤其是编码蛋白的基因。挑选靶区域时应避免选择与其他基因重叠的区域,且能影 响蛋白的功能结构域。
(2) 尽可能影响所有的转录本,敲除位点好在编码区的前 50%,但避免敲除 ATG 所在外显子或 ATG 之 前的外显子。
(3) 片段敲除的 sgRNA 设计在内含子上,这样能敲除整个外显子区,避免翻译出残留蛋白。敲除区域前后 序列尽量简单,方便后续的 PCR 鉴定。同时敲除的外显子编码序列之和为非 3 的倍数,使靶区域后面 的序列发生移码。另外,片段敲除所选定的敲除区域不要超过 10 Kb,超过 10 Kb 后编辑敲除效率会降低。
(4) 在设计 sgRNA 时要综合考虑候选编辑位点的序列、位置、正负链、GC 含量、潜在的脱靶位点等信息。例如靶点的 GC%尽量不要低于 40%,靶点序列 GC%偏高(50%~70%)有较高的打靶效率;靶点内不 要有连续 4 个以上的 T 碱基,避免形成 RNA Pol III 的转录终止信号等。
(5) 当敲除可能会导致细胞增殖非常缓慢或停止生长,或在细胞转染后的 cell pool 阶段检测效率呈显著下 降趋势,即可判定可能出现敲除致死的情况,建议用 RNAi。
(6) 由于存在部分强功能蛋白,即使表达下调了,仍然有较强的功能,如果 RNAi 始终做不出表型,但从有 关信息推测这个基因很大可能性会出表型,建议做 KO;另外,研究的目的基因处于非转录区域,或者 目的基因有很强的转录效率,也是无法用 RNAi 进行有效干扰的,则建议做 KO,且 KO 细胞更适合进 行回补实验。
(7) 后,敲低跟敲除不是二选一的关系。当我们用 RNAi 做了基因敲低,观察到细胞表型的变化后,仍然 可以进一步做敲除,让实验结论更加有说服性。
西安齐岳生物可提供的质粒构建服务包括:
(1)过表达质粒构建;
(2)诱导基因的定点突变;(单碱基突变,多碱基突变)
(3)构建基因截短体质粒;
(4)亚克隆质粒构建;
(5)通过慢病毒介导的基因过表达;
(6)通过构建shRNA慢病毒质粒实现基因的敲减
(7)通过CRISPR-Cas9技术实现基因敲除
请按照相关文献和相关安全规定进行操作,避免误用或滥用。如有侵权,可联系我们删除




 齐岳微信公众号
齐岳微信公众号 官方微信
官方微信 库存查询
库存查询